Sommaire des résultats du génotypage des souches de Clostridium difficile d’origine nosocomiale au Québec : 2017
La surveillance provinciale visant la caractérisation et la distribution des différents génotypes de Clostridium difficile dans les centres hospitaliers québécois a été initiée en 2005. Pour l’année 2017, la surveillance s’est échelonnée du 8 janvier au 27 mai 2017. Une prolongation de la surveillance a été nécessaire afin d’atteindre un nombre suffisant de souches à analyser, résultat d’une baisse marquée du nombre de cas de diarrhées associées à Clostridium difficile (DACD) d’acquisition nosocomiale depuis 2016. Tous les centres hospitaliers de la province étaient ainsi invités à fournir les 10 premiers échantillons de selles pour lesquels la recherche de toxines de C. difficile ou du gène de toxine s’est avérée positive dans leur laboratoire.
L'étude des souches
L’étude des souches pour l'année 2017 a porté sur 450 échantillons. Ces échantillons ont été obtenus de patients avec diarrhée à C. difficile d’acquisition nosocomiale. Les patients étaient suivis dans 68 centres hospitaliers répartis dans 16 régions sociosanitaires (RSS). Une souche de C. difficile a été isolée dans 417 des 450 (93 %) spécimens soumis. L’âge moyen des patients était de 72 ans, avec une médiane à 73 ans et un écart de 1 à 97 ans; 53 % étaient des femmes et 47 % des hommes.
La surveillance de laboratoire des souches de Clostridium difficile a été marquée par un changement notable en 2016 : la modification de la technique de typage. Le ribotypage a remplacé l’électrophorèse sur gel en champ pulsé (EGCP) qui a été utilisée pour les années de surveillance 2005-2015. La nomenclature des résultats de génotypage est donc modifiée. Ainsi, les souches NAP1 (North American Pulsotype 1) qui représentent le clone le plus prévalent au Québec, correspondaient selon la nomenclature de l’EGCP aux pulsovars A et A relié (exemple : A2-5). Selon la nomenclature du ribotypage, les souches NAP1 correspondent principalement au ribotype 027. Pour les autres ribotypes, il n’y a pas de correspondance entre les nomenclatures de l’EGCP et du ribotypage. La nouvelle nomenclature pour les résultats de ribotypage est numérique, où chaque combinaison de trois chiffres (exemple, 072) représente un ribotype particulier et donc, une souche différente. La concordance des génotypes NAP1 entre les pulsovars et les ribotypes est supportée par l’analyse des gènes de virulence (Persson et al., 2011). Rappelons que lors de la campagne de surveillance de 2015, cette analyse avait démontré que 100 % des souches de pulsovars A et A reliés (exemple : A2-5, A5) portaient la signature caractéristique de la souche NAP1, soit les gènes des toxines A, B et binaire et une délétion de 18 pb sur le régulateur négatif tcdC. À l’inverse, 96 % des souches autres que pulsovar A et A reliés contenaient aucune délétion sur le régulateur négatif tcdC et étaient négatives pour les gènes de la toxine binaire. Ce changement de technique de génotypage a été jugé nécessaire dans un but d’efficience, de précision et d’harmonisation avec les autres laboratoires effectuant de la surveillance, dont le Laboratoire national de microbiologie (Fawley et al., 2015).
Pour la surveillance 2017, le ribotypage, réalisé sur les 417 souches de C. difficile a permis d’identifier 76 ribotypes distincts pouvant être distribués en types épidémiques NAP1 à NAP12. Les résultats pour l’ensemble des centres hospitaliers ayant participé à la surveillance sont présentés au tableau 1 et une compilation provinciale au tableau 2 présente les différents profils NAP, leurs profils toxigéniques et les ribotypes associés. Les NAP1 représentent 29 % (120/417) des souches génotypées où 118 souches sont de ribotype 027, une souche de ribotype 075 et une souche de ribotype 171. L’ensemble des souches NAP1 porte les gènes caractéristiques, soient les gènes des toxines tcdA, tcdB, la délétion de 18 pb sur le régulateur négatif tcdC et la toxine binaire cdtA/cdtB. Les autres ribotypes d’importance sont le 106 (9 %) associé aux NAP11; 014 (8 %) et le 020 (7 %) tous deux associés aux NAP4. Leur profil toxigénique présente les gènes des toxines tcdA et tcdB sans la délétion sur le régulateur négatif (sauf 3 souches où une délétion de 18 pb a été observée) et sans la toxine binaire.
La figure 1 illustre le taux de NAP1 (Pulsovar A et A relié de 2005 à 2015 et ribotype 027, 075, 171, ns198 de 2016 à 2017). Elle permet d’observer la chute du taux de NAP1 dans nos établissements de santé depuis 2016, passant d’une moyenne de 62 % (écart de 58-69 %) de 2005 à 2015 à une proportion de 35 % pour 2016 et de 29 % pour 2017. La figure 2 présente l’évolution des différents profils NAP pour les surveillances 2016 et 2017 où la technique de ribotypage a été utilisée. Outre la diminution de la souche NAP1, il est possible d’observer une augmentation de la souche NAP4. Pour les années 2016 et 2017, la souche NAP4 représentait respectivement 15 et 18 % des souches isolées (n = 62; n = 75). L'augmentation de la NAP4 de 2016 à 2017 s'est principalement produite chez les ≥ 65. En effet, pour 2016 et 2017, le nombre de souches NAP4 représentait respectivement 22 et 23 souches chez les < 65 alors que pour les ≥ 65 ans, elles représentaient 40 et 52 souches. Le type NAP4 est présent dans la majorité des RSS du Québec, mais une augmentation plus marquée est retrouvée dans la RSS16 où le nombre de souches NAP4 chez les ≥ 65 est passé de 7 souches en 2016 à 15 souches en 2017. Une telle augmentation de la prévalence de la souche NAP4 a été rapportée ailleurs au Canada (Katz et al., 2018).
Conclusion
À la lumière de ces regroupements, il est possible d’observer qu’au cours des années de surveillance de laboratoire, la proportion de NAP1 au Québec a chuté subitement en 2016, passant d’une moyenne de 62 % de 2005 à 2015 à une proportion de 35 % pour 2016 et de 29 % pour 2017. De plus, l’utilisation de la technique de ribotypage depuis 2016 permet de documenter la présence des autres profils NAP, notamment la progression des souches de profils NAP4 dans nos établissements de soin.
Les laboratoires des centres hospitaliers ont reçu un rapport pour chaque prélèvement de selles soumis, incluant les résultats de culture, de gènes de virulence et de génotypage.
| RSS* | Centre hospitalier | NAP 1 | NAP 2 | NAP 4 | NAP 5 | NAP 6 | NAP 7 | NAP 9 | NAP 10 | NAP 11 | NAP 12 | Non classé† | Total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | Centre de SSS de Kamouraska | 1 | 1 | ||||||||||
| 1 | Centre de SSS de Rimouski-Neigette | 4 | 1 | 3 | 8 | ||||||||
| 2 | Centre de SSS Domaine-du-Roy | 1 | 4 | 5 | |||||||||
| 2 | Centre de SSS de Jonquière | 2 | 1 | 3 | |||||||||
| 2 | Centre de SSS de Lac-Saint-Jean-Est | 1 | 3 | 1 | 1 | 1 | 7 | ||||||
| 2 | Hôpital de Dolbeau-Mistassini | 1 | 1 | 2 | |||||||||
| 3 | CHU de Québec - Hôpital le CHUL | 1 | 2 | 3 | 6 | ||||||||
| 3 | CHU de Québec - Hôpital de l'Enfant-Jésus | 3 | 1 | 1 | 1 | 3 | 9 | ||||||
| 3 | CHU de Québec - Pavillon l'Hôtel-Dieu de Québec | 1 | 1 | 1 | 1 | 6 | 10 | ||||||
| 3 | CHU de Québec - Pavillon Saint-François-d’Assise | 2 | 2 | 1 | 1 | 3 | 9 | ||||||
| 3 | CHU de Québec - Hôpital du Saint-Sacrement | 1 | 1 | ||||||||||
| 3 | Institut Universitaire de Cardiologie et de Pneumologie de Québec | 2 | 1 | 1 | 4 | ||||||||
| 4 | Centre de SSS d'Arthabaska-et-de-l'Érable (Hôtel‑Dieu) | 1 | 1 | 2 | 4 | ||||||||
| 4 | Centre de SSS Drummond | 1 | 1 | 2 | |||||||||
| 4 | Centre de SSS de l'Énergie | 2 | 1 | 1 | 1 | 5 | |||||||
| 4 | Centre de SSS du haut-Saint-Maurice | 1 | 1 | 2 | |||||||||
| 4 | Centre de SSS de Trois-Rivières | 2 | 2 | 1 | 1 | 1 | 1 | 2 | 10 | ||||
| 5 | CHUS Fleurimont | 2 | 2 | 1 | 1 | 1 | 1 | 2 | 10 | ||||
| 5 | CHUS Hôtel-Dieu | 1 | 1 | 1 | 2 | 2 | 4 | 11 | |||||
| 5 | Centre de SSS de la Haute-Yamaska | 1 | 2 | 3 | |||||||||
| 5 | Centre de SSS de Memphrémagog | 1 | 2 | 3 | |||||||||
| 5 | Centre de SSS la Pommeraie | 2 | 2 | 4 | |||||||||
| 6 | Centre hospitalier de St-Mary | 3 | 4 | 3 | 10 | ||||||||
| 6 | CHUM Hôtel-Dieu | 1 | 1 | 2 | |||||||||
| 6 | CHUM Notre-Dame | 2 | 2 | 1 | 2 | 7 | |||||||
| 6 | CHUM St-Luc | 2 | 1 | 1 | 6 | 10 | |||||||
| 6 | CHU Sainte-Justine | 1 | 1 | 1 | 2 | 5 | |||||||
| 6 | Centre de SSS de l'Ouest-de-l'Île | 3 | 1 | 1 | 1 | 6 | |||||||
| 6 | Centre de SSS du Sud-Ouest-Verdun | 5 | 1 | 1 | 1 | 2 | 10 | ||||||
| 6 | CUSM - Hôpital Général de Montréal | 1 | 1 | 3 | 1 | 4 | 10 | ||||||
| 6 | CUSM - Hôpital Lachine | 1 | 1 | ||||||||||
| 6 | CUSM - Hôpital neurologique | 2 | 1 | 3 | |||||||||
| 6 | CUSM - Hôpital Royal-Victoria | 1 | 1 | 1 | 2 | 5 | 10 | ||||||
| 6 | Hôpital Général Juif Sir Mortimer B Davis | 4 | 1 | 1 | 2 | 2 | 10 | ||||||
| 6 | Hôpital Maisonneuve-Rosemont | 9 | 1 | 10 | |||||||||
| 6 | Hôpital du Sacré-Coeur de Montréal | 5 | 1 | 2 | 1 | 1 | 10 | ||||||
| 6 | Hôpital Fleury | 1 | 2 | 2 | 2 | 7 | |||||||
| 6 | Hôpital Jean-Talon | 3 | 1 | 1 | 5 | ||||||||
| 6 | Hôpital Santa Cabrini | 7 | 1 | 1 | 1 | 10 | |||||||
| 6 | Institut de Cardiologie de Montréal | 4 | 1 | 5 | |||||||||
| 7 | Centre de SSS de Gatineau (Hôpital de Gatineau) | 1 | 1 | 1 | 1 | 1 | 3 | 8 | |||||
| 7 | Centre de SSS de Gatineau (Hôpital de Hull) | 2 | 2 | 2 | 6 | ||||||||
| 7 | Centre de SSS de la Vallée-de-la-Gatineau | 1 | 1 | ||||||||||
| 8 | Centre de SSS de Rouyn-Noranda | 1 | 1 | 1 | 1 | 1 | 5 | ||||||
| 8 | Centre de SSS de La Vallée-de-L'Or | 2 | 2 | 1 | 1 | 1 | 7 | ||||||
| 9 | Centre de SSS de la Minganie | 1 | 1 | ||||||||||
| 9 | Centre de SSS de Sept-Îles | 1 | 1 | 1 | 3 | ||||||||
| 11 | Centre de SSS de la Baie-des-Chaleurs | 3 | 2 | 5 | |||||||||
| 11 | Centre de SSS des Îles | 1 | 1 | ||||||||||
| 12 | Centre de SSSS Alphonse-Desjardins (Hôtel-Dieu Lévis) | 1 | 4 | 1 | 1 | 3 | 10 | ||||||
| 12 | Centre de SSS de Beauce (St-Georges) | 3 | 2 | 1 | 2 | 1 | 9 | ||||||
| 12 | Centre de SSS de la Région de Thetford (Hôpital Thetford-Mines) | 1 | 1 | 1 | 2 | 5 | |||||||
| 12 | Hôpital Hôtel Dieu Montmagny | 1 | 1 | 2 | |||||||||
| 13 | Centre de SSS de Laval (Cite de la Santé) | 4 | 1 | 4 | 1 | 10 | |||||||
| 14 | Centre de SSS du Nord de Lanaudière | 6 | 1 | 2 | 1 | 10 | |||||||
| 14 | Centre de SSS du Sud de Lanaudière | 1 | 1 | ||||||||||
| 15 | CSSS d'Antoine-Labelle Hôpital de Mont-Laurier | 1 | 1 | 2 | |||||||||
| 15 | Centre de SSS du Lac-des-Deux-Montagnes | 2 | 2 | 1 | 1 | 4 | 10 | ||||||
| 15 | Centre de SSS de Saint-Jérôme | 8 | 2 | 10 | |||||||||
| 15 | Centre de SSS des Sommets | 5 | 1 | 1 | 1 | 1 | 9 | ||||||
| 16 | Centre de SSS Champlain-Charles-Le Moyne | 1 | 5 | 1 | 2 | 9 | |||||||
| 16 | CSSS Haut-Richelieu-Rouville | 2 | 3 | 1 | 2 | 2 | 10 | ||||||
| 16 | Centre de SSS Jardins-Roussillon | 1 | 1 | 2 | 1 | 2 | 2 | 9 | |||||
| 16 | Centre de SSS Pierre-Boucher | 2 | 3 | 2 | 7 | ||||||||
| 16 | Centre de SSS Pierre-de Saurel | 2 | 2 | ||||||||||
| 16 | CISSS de la Montérégie-Est (Hôpital Honore-Mercier) | 3 | 1 | 1 | 5 | 10 | |||||||
| 16 | Centre de SSS du Suroît | 2 | 1 | 1 | 4 | ||||||||
| 18 | Conseil Cri de SSS de la Baie-James | 1 | 1 | ||||||||||
*Région sociosanitaire
† Non classé : regroupent les ribotypes qui ne sont pas associés à un profil NAP
| Classement NAP | Profils toxigéniques | Ribotype | Nb de souches |
|---|---|---|---|
| NAP1 | positif tcdA et tcdB; tcdC délétion 18 pb; positif toxine binaire cdtA/cdtB | 027, 075, 171, ns198 | 262 |
| NAP2 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 001 / 072 | 28 |
| NAP4 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 006, 014, 020, 076, 096, 154, 207, 221, ns 178, ns186 | 137 |
| NAP5 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 53 | 1 |
| NAP6 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 2 | 32 |
| NAP7 | positif tcdA et tcdB; tcdC délétion 39 pb; positif toxine binaire cdtA/cdtB | 078, 126, ns163 | 12 |
| NAP9 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 17 | 9 |
| NAP10 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 003, 011, 057, 070, 137, ns199 | 39 |
| NAP11 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 103, 106, 351 | 86 |
| NAP12 | positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB | 015, 087 | 35 |
| positif tcdA et tcdB; tcdC délétion 18 pb; négatif toxine binaire cdtA/cdtB | |||
| Non classé | négatif tcdA, tcdB et tcdC; négatif toxine binaire cdtA/cdtB | 005, 090, 010, 012, 013, 018, 019, 023, 029, 039, 043, 045, 046, 050, 054, 056, 080, 081, 084, 097, 098, 104, 153, 176, 293, 305, 328, 385, 404, 511, ns38, ns85, ns98, ns99, ns107, ns123, ns131, ns136, ns145, ns165, ns179, ns180, ns181, ns185, ns187, ns190, ns192, ns207, ns214, ns215, ns216, ns226, ns264, ns265, ns266, ns274, Qc1, Qc2, Qc3, Qc4, Qc5, Qc6 | 180 |
| positif tcdA et tcdB; tcdC délétion 18 pb; positif toxine binaire cdtA/cdtB | |||
| positif tcdA et tcdB; tcdC délétion 39 pb; positif toxine binaire cdtA/cdtB | |||
| positif tcdA et tcdB; tcdC délétion 54 pb; positif toxine binaire cdtA/cdtB | |||
| positif tcdA et tcdB; tcdC sans délétion; positif toxine binaire cdtA/cdtB | |||
| positif tcdA et tcdB; tcdC sans délétion; négatif toxine binaire cdtA/cdtB |
Figure 1 - Répartition des souches de C. difficile (%) de profils NAP1 et autre que NAP1 recensées au cours des surveillances de laboratoire provinciales des DACD
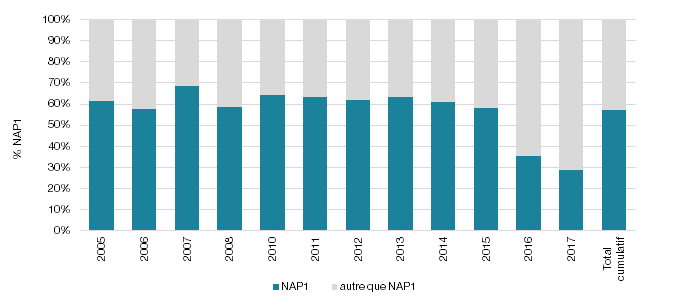
Pour les années de surveillance 2005 à 2015, les % NAP1 incluent les pulsovars A et A relié (ex. : pulsovar A, A2-5, A5).
Pour l’année 2016-2017, le % NAP1 est déterminé par ribotypage et supporté par le profil toxigénique (027, 075, 171, ns198).
N = 5048 souches analysées.
Figure 2 - Répartition des souches de C. difficile (n) selon leur profil NAP recensées au cours des surveillances de laboratoire provinciales des DACD de 2016 et 2017 (n = 821)
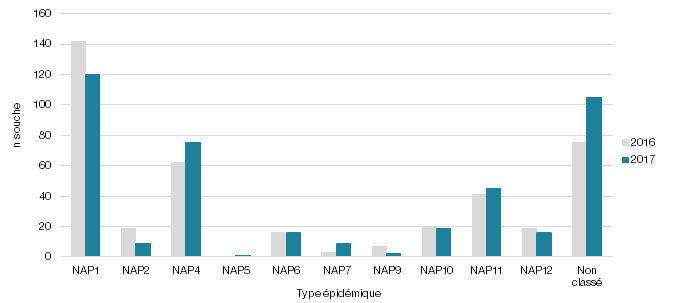
Références
- Fawley, W.N., Knetsch, C.W., MacCannell, D.R., Harmanus, C., Du, T., Mulvey, M.R., Paulick, A., Anderson, L., Kuijper, E.J., Wilcox, M.H., 2015. Development and Validation of an Internationally-Standardized, High-Resolution Capillary Gel-Based Electrophoresis PCR-Ribotyping Protocol for Clostridium difficile. PLOS ONE 10, e0118150. https://doi.org/10.1371/journal.pone.0118150
- Katz, K.C., Golding, G.R., Choi, K.B., Pelude, L., Amaratunga, K.R., Taljaard, M., Alexandre, S., Collet, J.C., Davis, I., Du, T., Evans, G.A., Frenette, C., Gravel, D., Hota, S., Kibsey, P., Langley, J.M., Lee, B.E., Lemieux, C., Longtin, Y., Mertz, D., Mieusement, L.M.D., Minion, J., Moore, D.L., Mulvey, M.R., Richardson, S., Science, M., Simor, A.E., Stagg, P., Suh, K.N., Taylor, G., Wong, A., Thampi, N., 2018. The evolving epidemiology of Clostridium difficile infection in Canadian hospitals during a postepidemic period (2009–2015). Can. Med. Assoc. J. 190, E758–E765. https://doi.org/10.1503/cmaj.180013
- Persson, S., Jensen, J.N., Olsen, K.E.P., 2011. Multiplex PCR Method for Detection of Clostridium difficile tcdA, tcdB, cdtA, and cdtB and Internal In-Frame Deletion of tcdC. J. Clin. Microbiol. 49, 4299–4300. https://doi.org/10.1128/JCM.05161-11

